code
other-other文章的研究过程和结论,每段代码的作用和结果介绍
来自nature文章Improved prediction of immune checkpoint blockade efficacy across multiple cancer types
具体代码实现详见我的GitHub仓库
研究过程
文章的主要目的:开发一个机器学习模型,通过全面整合与免疫疗法疗效相关的多种生物学特征,准确预测患者免疫治疗反应的可能性,同时也可以间接预测生存状态(预后)
GridSearch and RandomForestClassifier
这部分主要做了两件事:
-
探究哪些因素对ICB反应的影响最大
-
选出其中的16/11个因素,分别构建了
RF16和RF11模型
作者首先按癌症类型将数据集随机分成训练组和验证组(测试组),训练组有80%的样本(n=1184)、验证组有20%的样本(n=295)
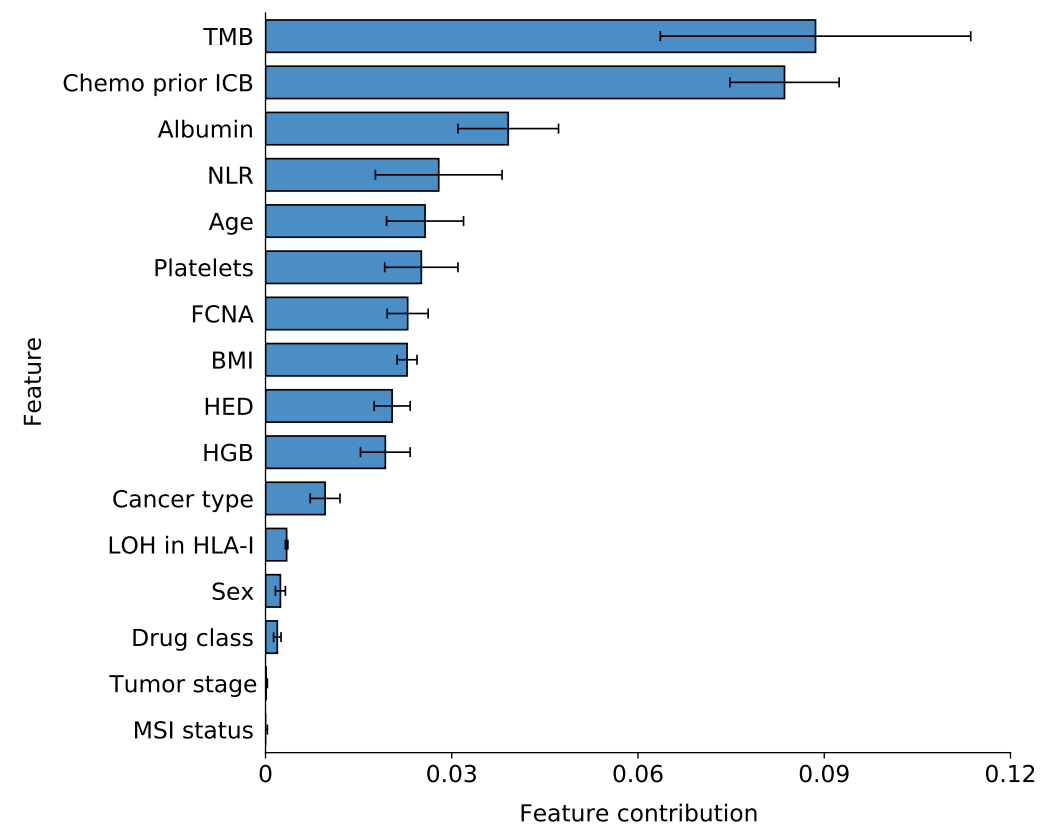
之后作者先选了16个因素,包括癌症类型、Albumin、HED、TMB、FCNA、BMI、NLR、Platelets、HGB、Stage、Age、Drug、Chemo_before_IO、HLA_LOH、MSI、Sex,基于它们使用GridSearch方法构建了一个模型RF16,之后又用PermutationImportance方法评估每个因素对模型的贡献(影响)
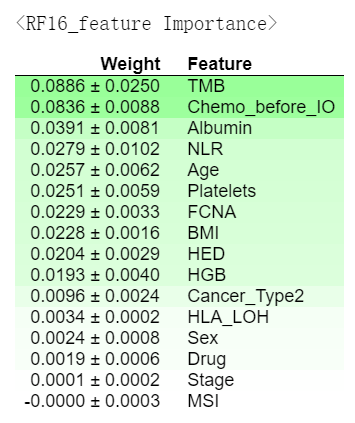
如图所示,Weight就是贡献大小
其中肿瘤突变负荷TMB的贡献最大,这与许多其它研究的结果一致;化疗史Chemo_before_IO对ICB反应的影响与TMB相似;MSI状态并未被模型选为主要预测因子之一,可能是因为它与TMB密切相关
之后作者为了评估将癌症类型、Chemo_before_IO以及血液标志物(Platelets、HGB、Albumin)与其他因素相结合的预测能力,又选了11个因素,包括HED、TMB、FCNA、BMI、NLR、Stage、Age、Drug、HLA_LOH、MSI、Sex(注意这里不是上面RF16中影响最大的11个因素),目的是确定以前未广泛使用的其他因素(指影响较小的因素?)来预测ICB
建模方法同上,结果如下
两种模型的准确性:
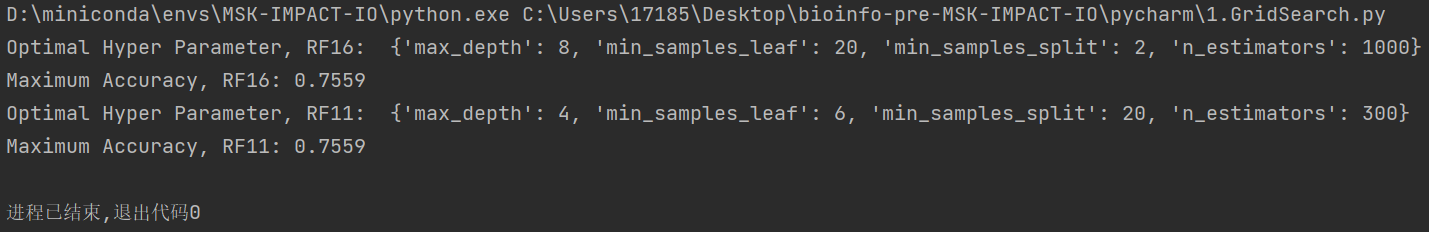
RF16模型的最高准确率为0.7559,RF16模型的最高准确率为0.7576(由于我这里只运行了一次,不一定是最高值)
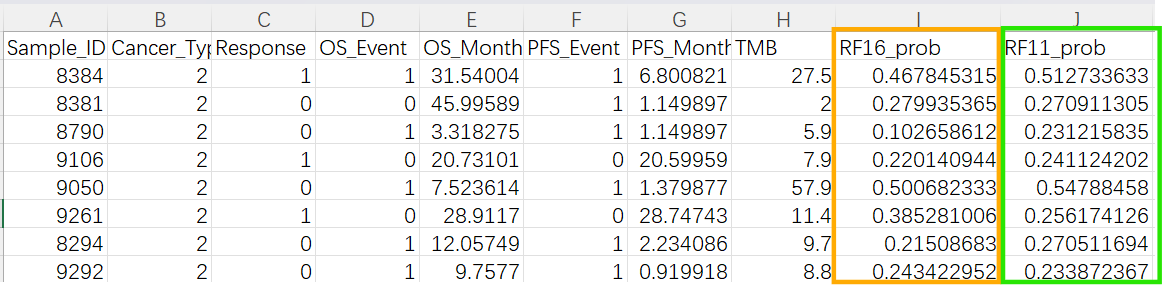
最后得到两个txt文件(Test_RF_Prob.txt和Training_RF_Prob.txt),其中的RF16_prob和RF11_prob列,分别是验证组和训练组中的样本 使用两个模型(RF16/RF11) 预测的得分(对ICB免疫疗法有反应的概率)
作者在这部分画了两组图:
-
统计各类癌症NR和R的人数
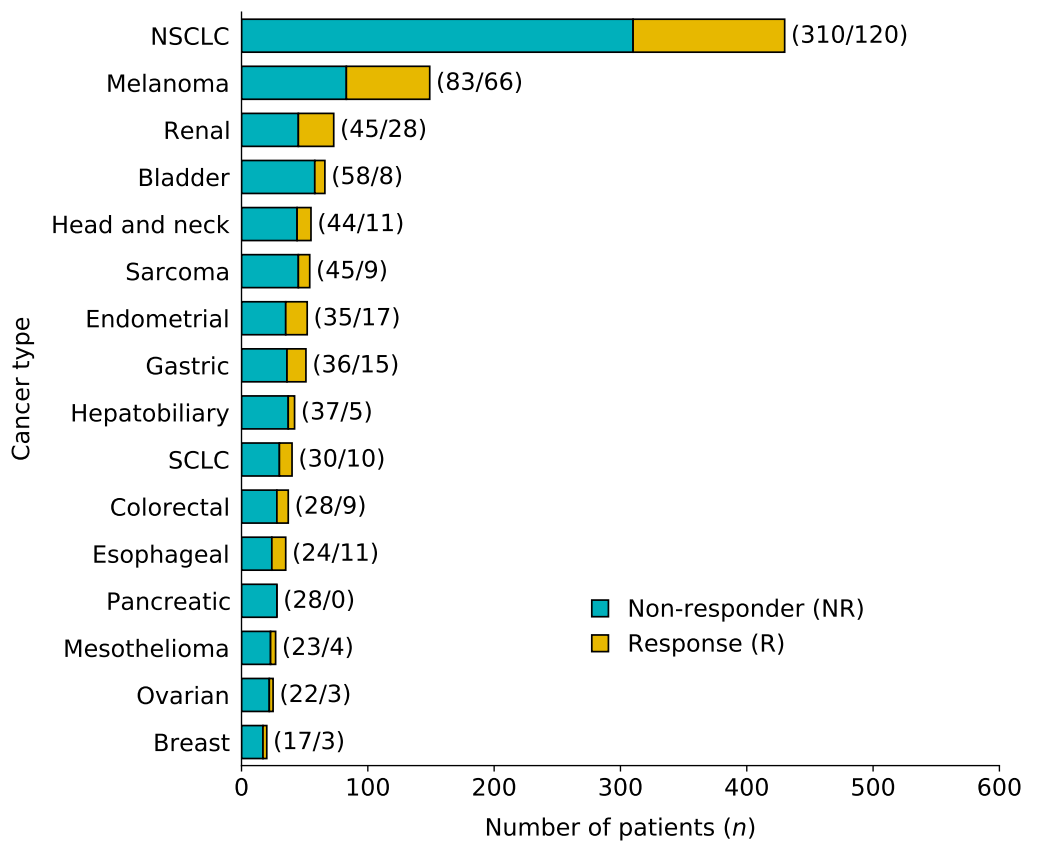
-
展示RF16模型中各因素的影响大小
ROC and PRC
对于训练组数据,作者根据样本的癌症种类,对上面得到的数据又进行了4次“分组”,分别是
-
Pan-cancer–包含全部癌症种类(泛癌) -
NSCLC–只包含非小细胞肺癌 -
Melanoma–只包含黑色素瘤 -
Others–除了非小细胞肺癌和黑色素瘤的其它癌症
根据这四组数据,作者使用RF16模型建了4个子模型,分别是泛癌模型和3个癌症特异性模型
注意:这里所说的“泛癌模型”和“癌症特异性模型”,都是基于上面的RF16_prob和RF11_prob打分,只是选取的最佳阈值不同(泛癌模型是对全部样本计算,只选取一个阈值;而癌症特异性模型是将不同癌症的样本分开来计算,每种癌症都有一个单独的阈值)
对每个模型都画了ROC和PRC曲线,并计算了各自的最佳阈值,结果保存在Pan_Thresholds.txt(泛癌模型)和Thresholds.txt(癌症特异性模型)中
以“训练组–泛癌”为例:
-
ROC-PRC曲线及其具体数值:
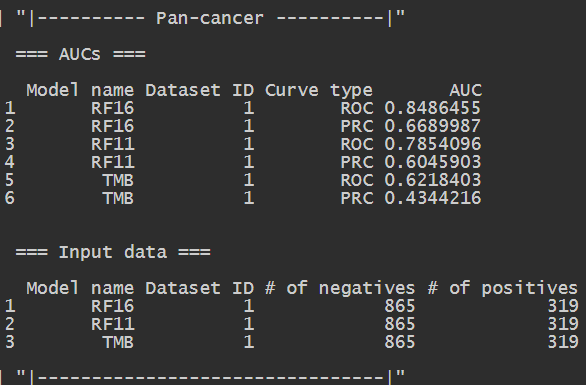
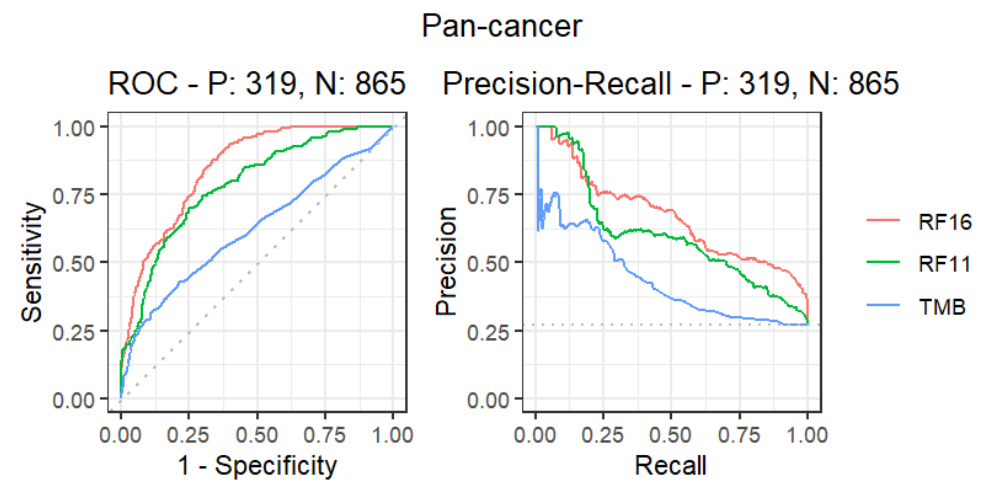
可以看到RF16模型在ROC中最靠左上角,在PRC中最平滑且靠上,对患者是否有应答的预测能力最好
-
在ROC曲线上取最佳阈值:
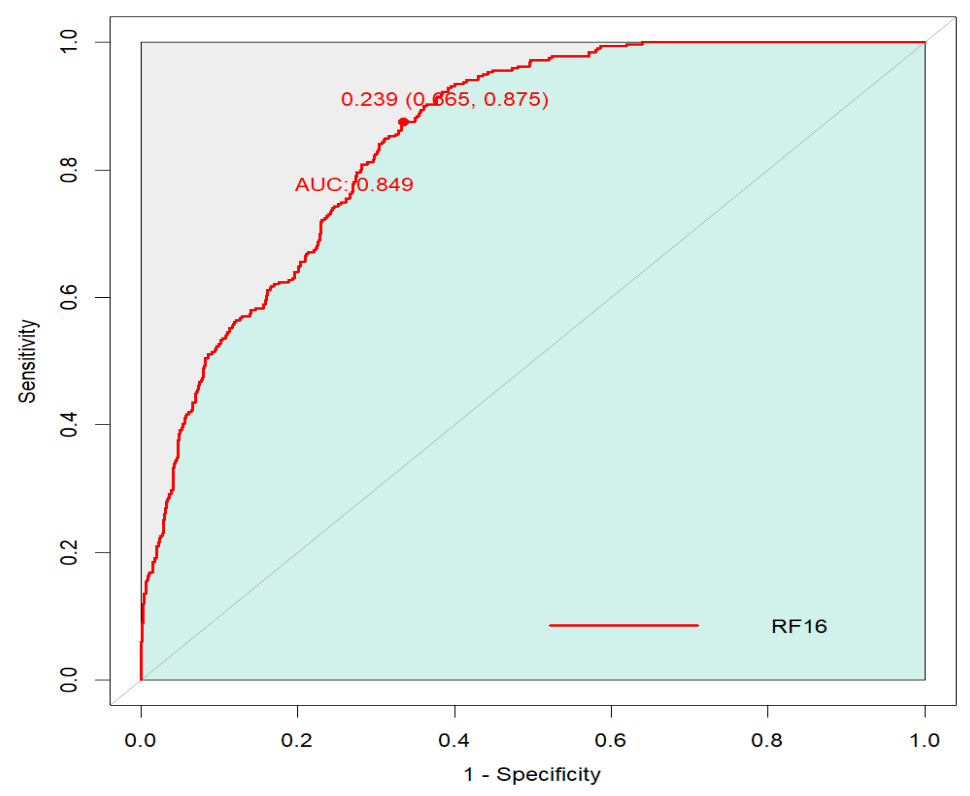
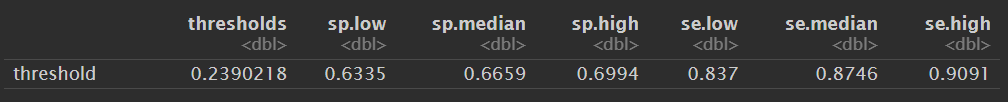
曲线上标的那个点就是最佳阈值点,点上标注的是
最佳阈值 (特异度, 敏感度)
所有的最佳阈值结果:
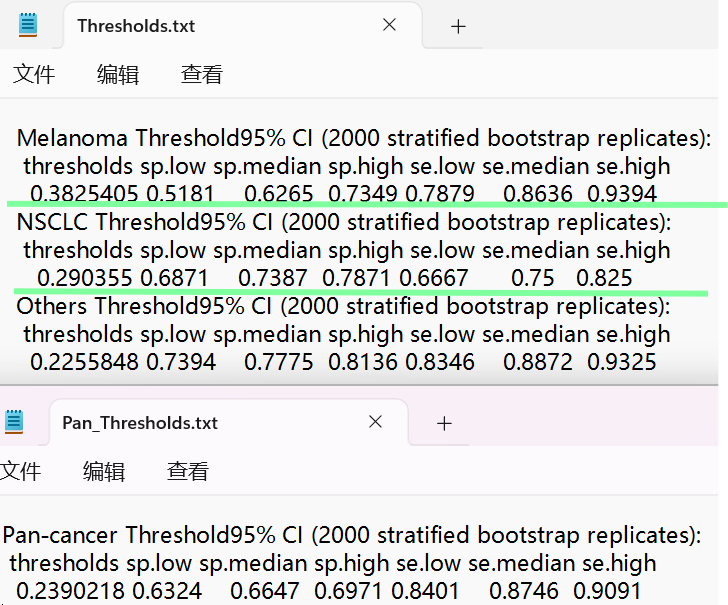
对于验证组,分组方式同训练组,但只画了ROC和PRC曲线,没有计算最佳阈值
作者在这部分画了两组图:
-
各模型(
RF16/RF11/TMB)对泛癌和3种癌症的ROC曲线:-
验证组:
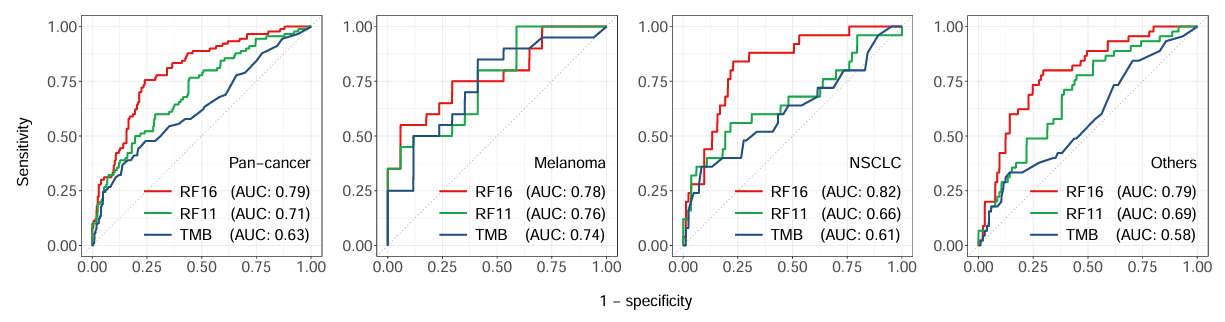
-
训练组:
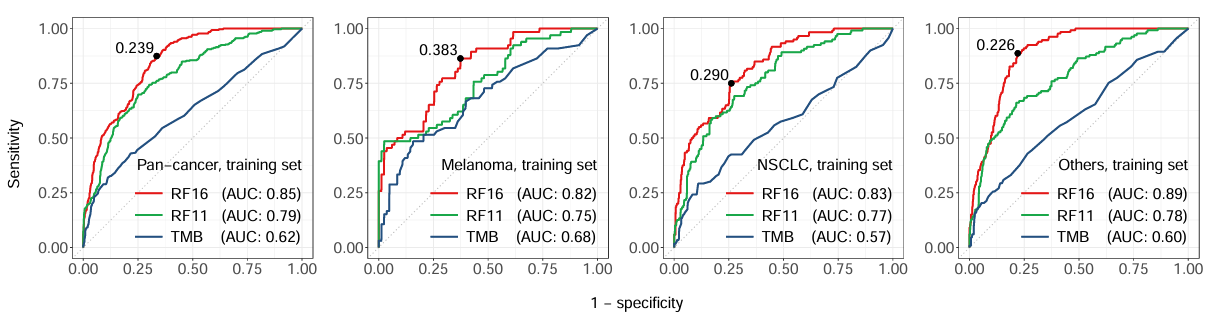
可以看到验证组和训练组中,
RF16模型对各种癌症预测的AUC均最高 -
-
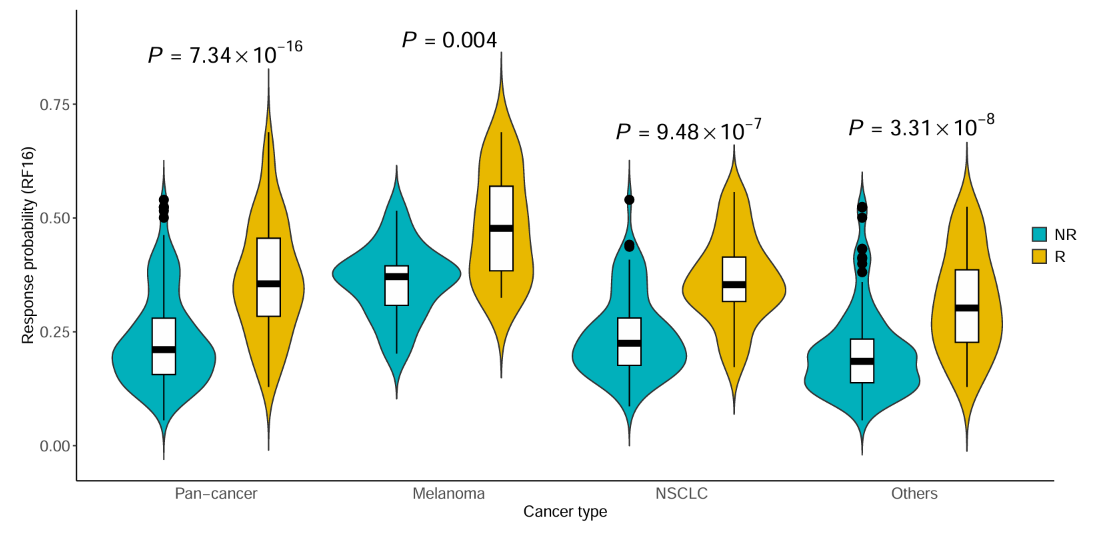
小提琴图:将数据按NR和R、癌症种类的不同进行分组,分别比较RF16预测结果和TMB
RF16_prob–癌症种类:
- log2(TMB+1)–癌症种类:
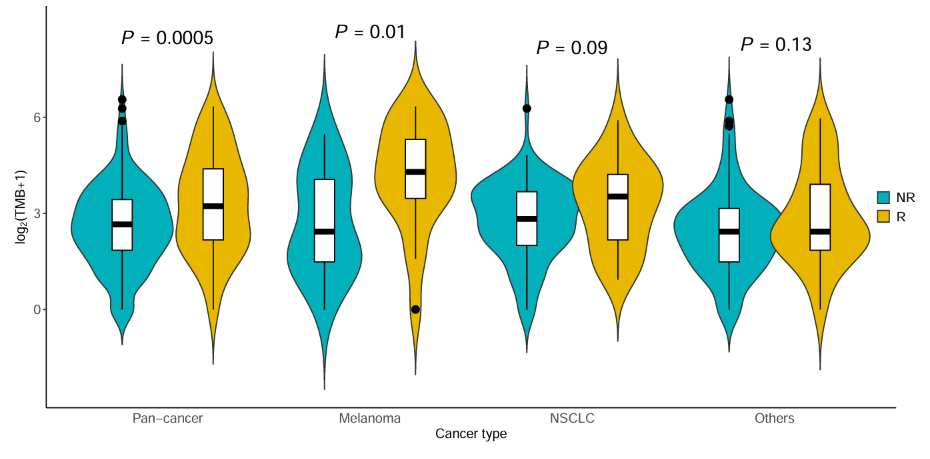
可以看到,在4种癌症中,RF16模型预测得分的组间差异都明显大于TMB的
Evaluate Performance
在这一部分(5-7的python代码)中,作者干了2件事
-
在最早的
Test_RF_Prob.txt和Training_RF_Prob.txt中新添一列或两列,标明模型或者TMB预测各样本是R还是NR-
如果RF16模型给出的预测值(
RF16_prob)≥最佳阈值,就说明模型预测该样本为R(应答者),否则为NR(非应答者)注:如果是特异性模型,就根据每个样本的癌症种类选择对应的癌症模型阈值。比如这个样本是Melanoma,就找Melanoma模型的最佳阈值,将
RF16_prob与这个阈值比较 -
如果TMB≥10,则根据TMB的预测结果为R,否则为NR
训练组的泛癌模型数据只统计了RF16模型预测结果
Training_RF_Prob_Pan_Predicted.txt,训练组和验证组的癌症特异性模型数据统计了RF16模型和TMB预测结果Training_RF_Prob_Predicted.txt/Test_RF_Prob_Predicted.txt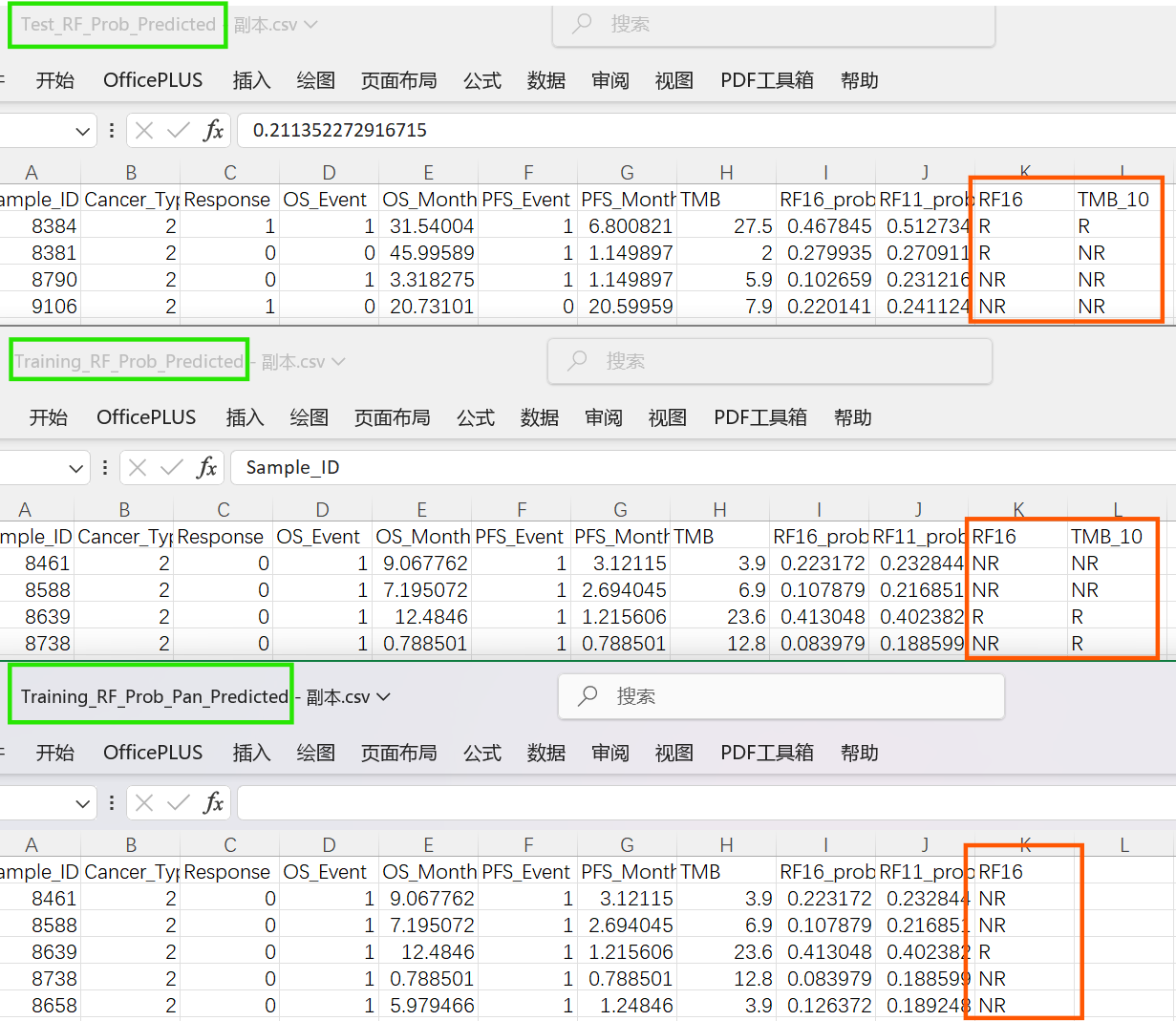
注:因为后面有一张图还需要RF11模型的预测结果,所以代码中是添加了2/3列(多了RF11的)
-
-
根据上面的预测结果,计算并输出各模型对各种癌症的预测灵敏度、特异性、准确率、阳/阴性预测值
其实就是统计上面图中5组预测结果(5个红框),与真实值
Response相对比,看看预测对了没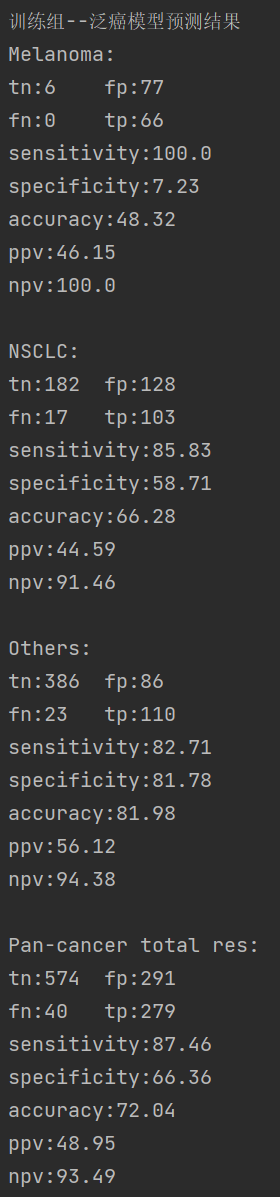
类似的还有4个输出,除了上面的“训练组–泛癌模型预测结果”,还有“训练组–特异性模型预测结果”、“训练组–TMB预测结果”、“验证组–特异性模型预测结果”、“验证组–TMB预测结果”
作者在这部分画了两组图:
-
混淆矩阵:展示验证组中,
RF16和TMB模型对各种癌症的预测结果(TP、TN、FP、FN)。这里以这两个模型对泛癌的预测结果为例: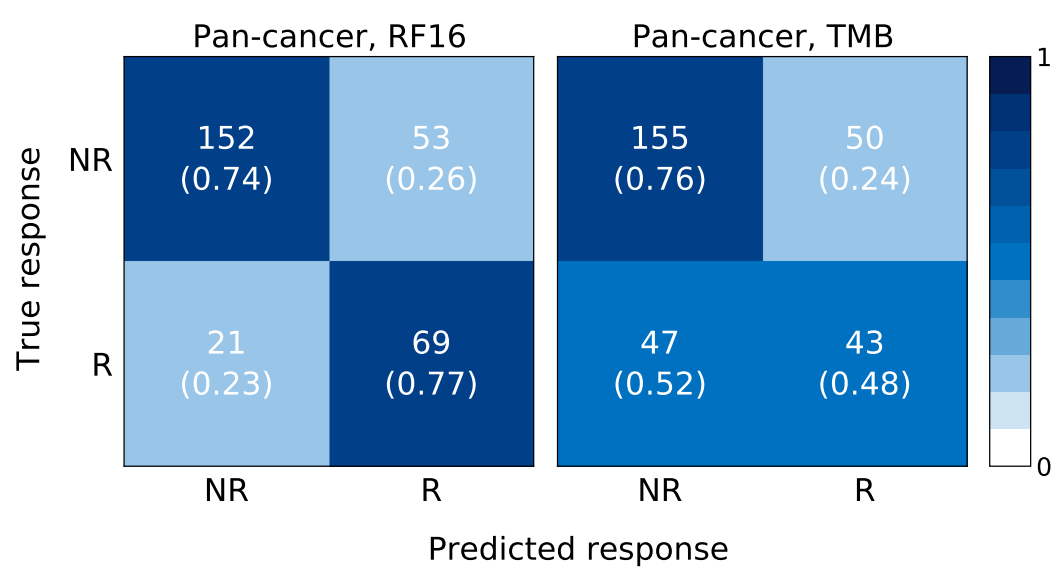
-
柱状图:展示验证组中,各模型对各种癌症的预测结果(灵敏度、特异性、准确率、阳/阴性预测值)
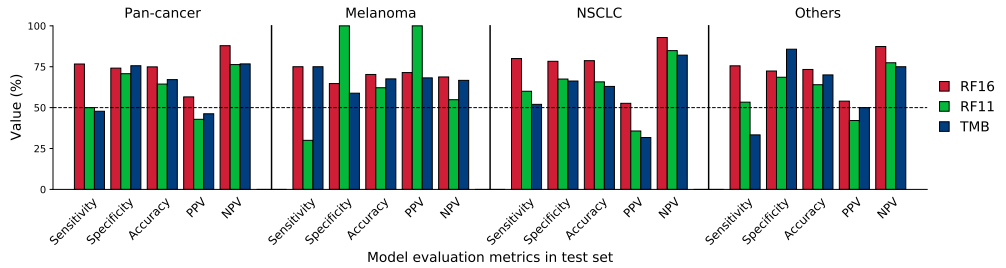
横坐标就是5种预测结果,纵坐标是结果值(以百分比为单位),不同颜色代表不同模型,共分成了4大组(每组间以竖线分隔)
文章最前面概述部分也画了一个混淆矩阵,可能使用的是测试组泛癌的预测结果
Brier score
进行生存分析,探究模型预测得分(RF16_prob/RF11_prob/TMB)与生存状态OS的关系
也是将训练组和验证组分开来,每组都进行泛癌、Melanoma、NSCLC、Others共4组分析,计算了Brier score并绘制了Prediction error curves图
以“训练组–Pan-cancer”为例:
-
Brier score分析结果:
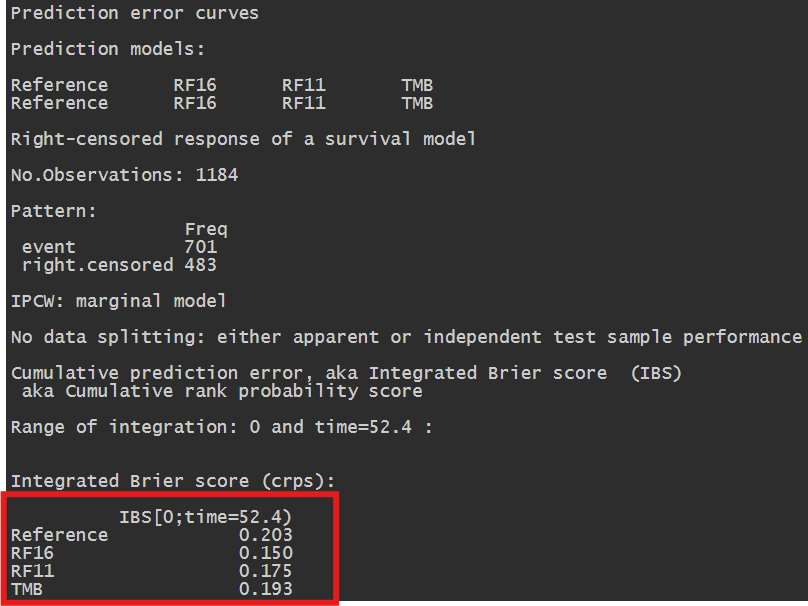
红框内的就是各模型关于生存状态的总Brier score
Reference:画图函数提供的,使用marginal Kaplan-Meier方法建模进行预测的结果
-
Prediction error curves图:
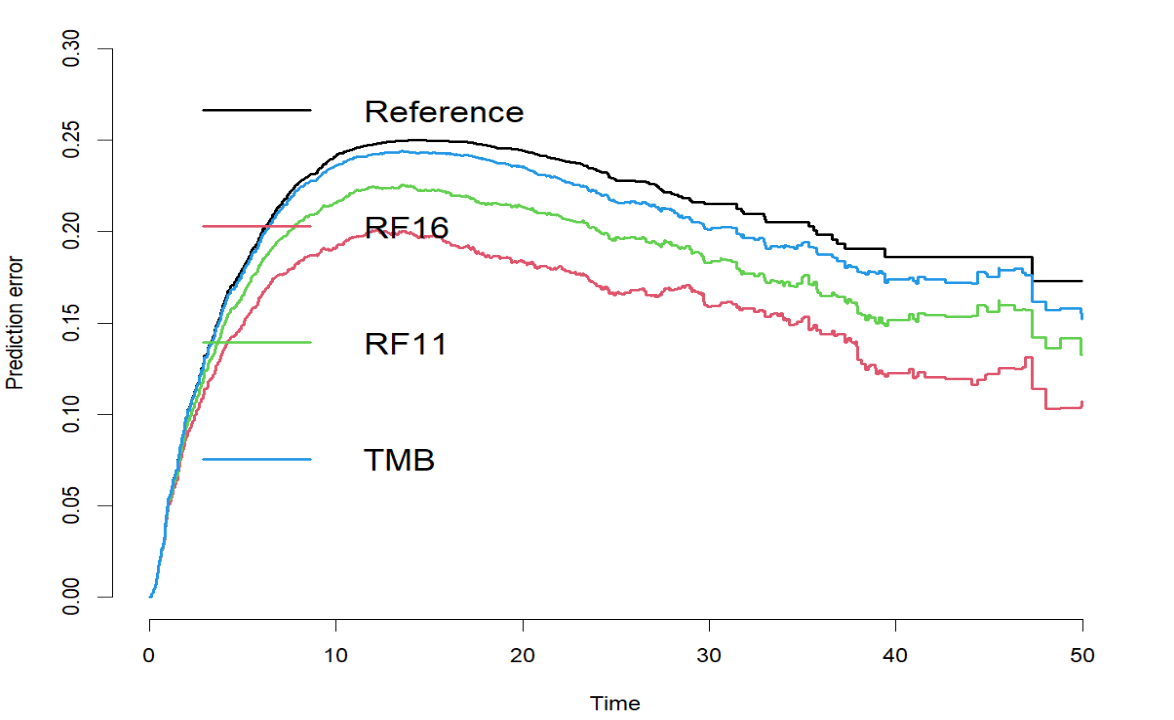
该图展示了模型对不同生存时间的患者的预测能力,纵坐标是Brier score,因此曲线越靠下预测效果越好。可以看到RF16(红色线)对生存状态的预测能力几乎也是最好的
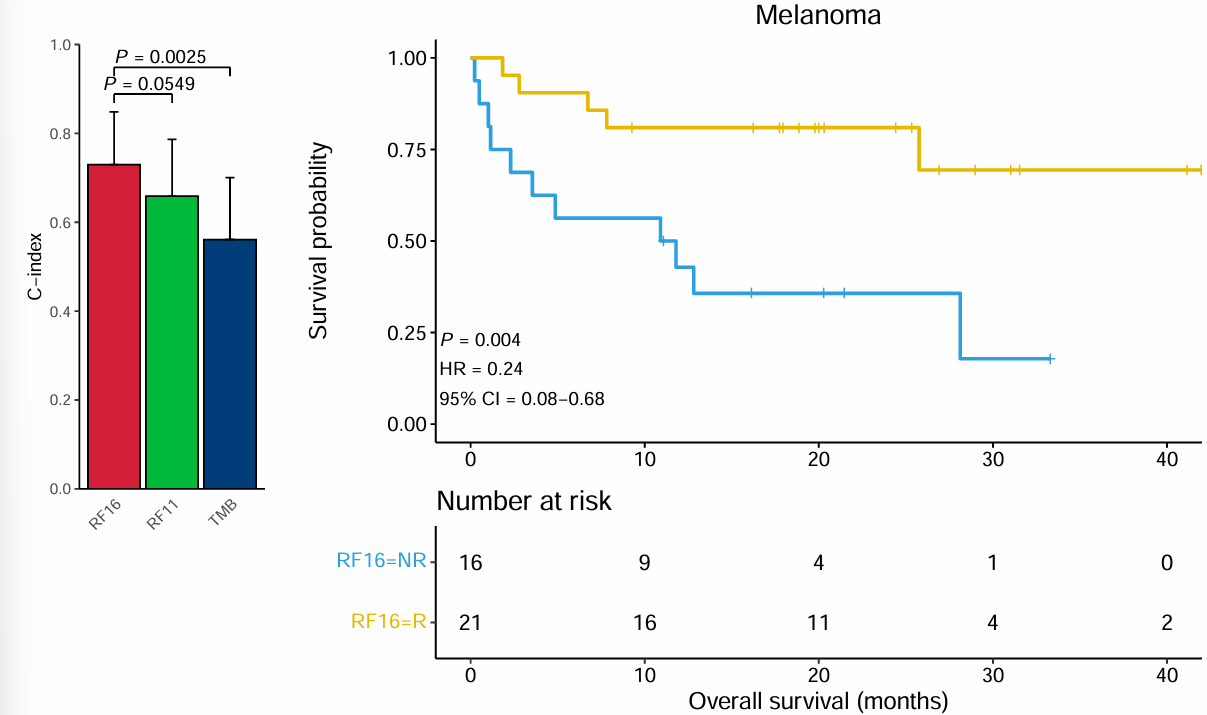
C-index
计算了训练组和验证组中,RF16_prob/RF11_prob/TMB这3个模型对于两种生存状态(OS和PFS)的预测能力,也是分成泛癌、Melanoma、NSCLC、Others共4组,并比较了每个模型c-index值的差异
以测试组–Melanoma–OS为例:
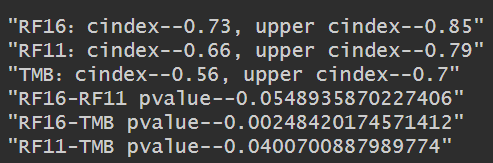
upper_cindex是误差,在下图中作为误差棒的上界
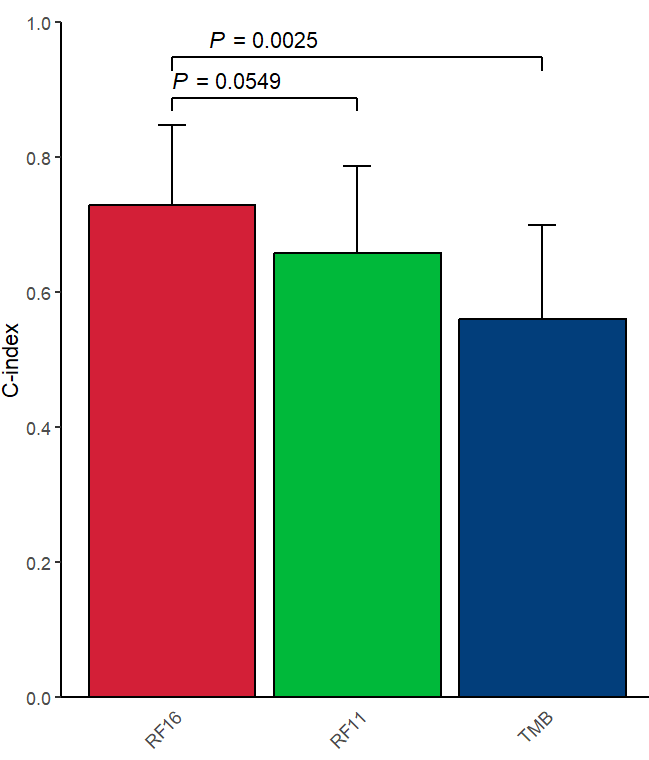
可以看到RF16的cindex值较高,且与另两组间p值基本都<0.05,说明RF16显著优于另两组模型
Survival
进行生存分析:根据RF16模型的预测结果,将样本分为R和NR两组,计算这两组生存状态的差异
以测试组–Melanoma–OS为例:
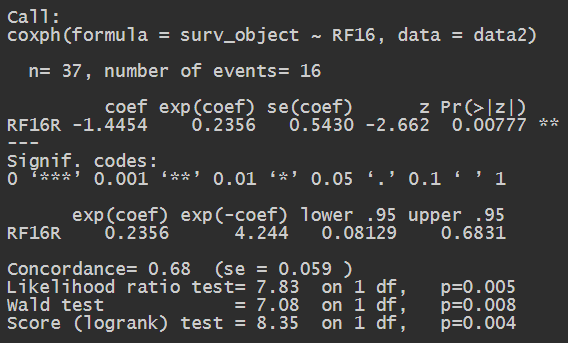
绘制survplot图:
横坐标是时间,下面图的纵坐标是存活人数,即在两组中,在对应时间有多少人还存活;上面图的纵坐标是生存概率,根据下面图的存活人数/总人数计算得到
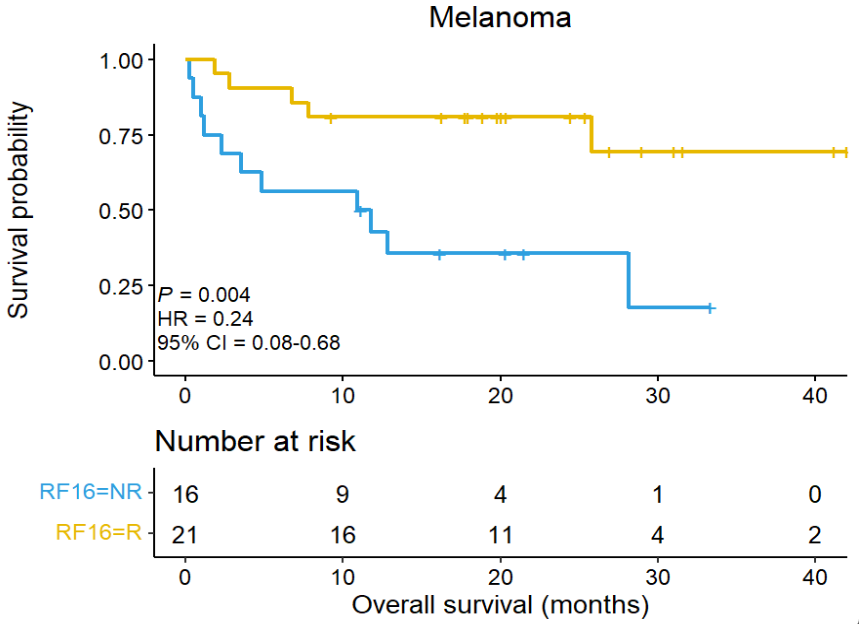
可以看到R(有应答)组的生存概率明显高于NR(无应答)组,HR值为0.24(远小于1),p值<0.05且CI没有跨过“1”这个点(说明得到的HR有统计学意义),说明模型的分组能较准确地预测生存状态
在作者提供的代码中,对于训练组和验证组这2组,作者用RF16/TMB这2个模型都画了图,每组都分为泛癌+3种癌症共4种,生存状态也分为OS和PFS2种,因此共画了2*2*4*2=32张图,但论文实际呈现的图只有验证组的RF16模型的8张图,因此我的代码中也只画了这8张(C-index也是)
作者将cindex和生存分析的结果画到了一张图中,还是以测试组–Melanoma–OS为例:
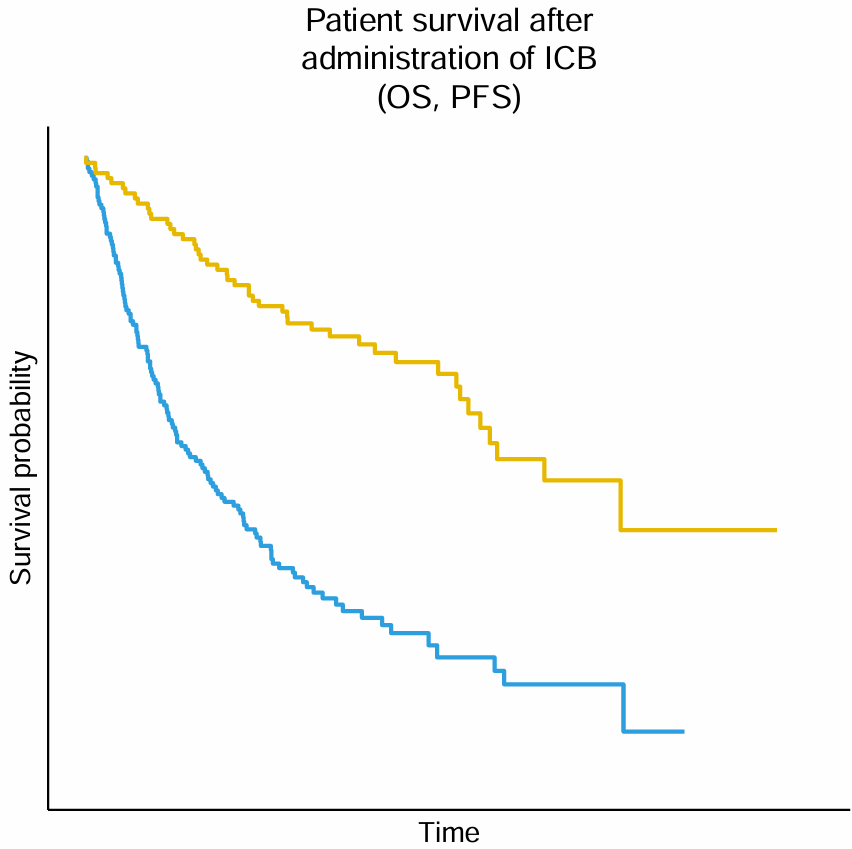
文章最前面概述部分也画了一个生存曲线,我这里用的验证组的OS,原图应该用的不是作者提供的数据集(与概述部分的横向堆叠柱状图一样)
结论
RF16模型:
-
选用因素:
-
血液成分测量:白蛋白、血红蛋白、血小板水平,中性粒细胞与淋巴细胞比值
-
肿瘤相关信息:肿瘤突变负荷,拷贝数改变分数,微卫星不稳定性
-
患者基本信息:BMI,年龄,性别
-
患者基因信息:HLA-I进化差异、杂合性缺失状态
-
患者其它信息:肿瘤类型和分期、使用的免疫治疗药物,免疫治疗前是否接受化疗
-
-
评估依据:免疫治疗是否有应答,OS和PFS生存状态
作者在文中的总结:
-
该机器学习模型可以在免疫治疗之前高精度地预测ICB治疗反应、OS和PFS
-
准确预测ICB反应需要一个综合模型,该模型结合了患者基本信息(BMI和年龄性别)、基因组特征、临床信息、体现患者整体健康状况的血液标志物。每个特征都可以较容易地从肿瘤组织DNA测序、血常规检查中得到
-
为理解和量化免疫疗法的对不同患者的疗效提供了更精细的方法
-
表明多个生物因素的非线性组合不同程度地影响了ICB疗效
-
局限性:没有转录组数据和tumor PD-L1 staining(肿瘤组织染色),这些是评估肿瘤微环境的重要信息
作者在文中的其它补充:
-
研究使用的患者数据库规模有限,不一定代表全球目标人群,需要在临床试验的背景下对其他更多患者进行测试
-
肿瘤免疫微环境的分子特征、微生物组组成、T细胞受体库的多样性、特异性肿瘤基因组改变(例如与DNA损伤和修复相关的基因突变、与ICB耐药相关的基因突变)、转录组数据,这些数据可能会进一步提高预测性能,这种定量模型将在精准免疫肿瘤学领域对改善患者预后具有重要意义